Cell Reports | 生信分析 | 全基因组识别和分析人类癌症的预后特征
中文题目:全基因组识别和分析人类癌症的预后特征
期刊:Cell Reports
IF:9.423
DOI: https://doi.org/10.1016/j.celrep.2022.110569
2.拷贝数改变和转录本比突变更具预后意义
3.癌基因和成功的癌症药物靶点很少有预后作用
4.创建了一个门户网站以促进与结果相关的生物标志物的分析
他们确定了数千个预后生物标志物,并揭示了基因拷贝数改变与患者死亡之间的显著关联。
可以挖掘这一综合资源来探寻最有可能揭示患者疾病进展的生物标志物。
基因表达分析、DNA 测序、拷贝数测定和其他基因组技术在临床环境中的广泛应用,提高了分子标记的开发以改善风险评估。
最近的一系列研究强调,拷贝数改变 (CNA) 也传达了重要的预后信息,增加的 CNA 负担通常与疾病复发和转移传播有关。
然而,现有研究存在一些局限性:
(1)已发表的分析主要集中在单一癌症和/或基因组数据类型。 尚未报道跨癌症类型和基因组平台的预后生物标志物的全面比较。
(2)生物标志物研究可能会受到“文件抽屉问题”(发表偏倚)的影响,如发现新的生物标志物更有可能被发表,而负面结果可能最终会出现在文件抽屉中。
为了提高我们识别最具侵袭性的恶性肿瘤的能力,研究使用来自 10,884 名患者的基因表达、拷贝数、甲基化和突变数据构建了全基因组生存模型。
TCGA 队列中的生存分析→分析方法的选择→整体分析策略→Kaplan-Meier分析→基因本体分析→其他工具和资源
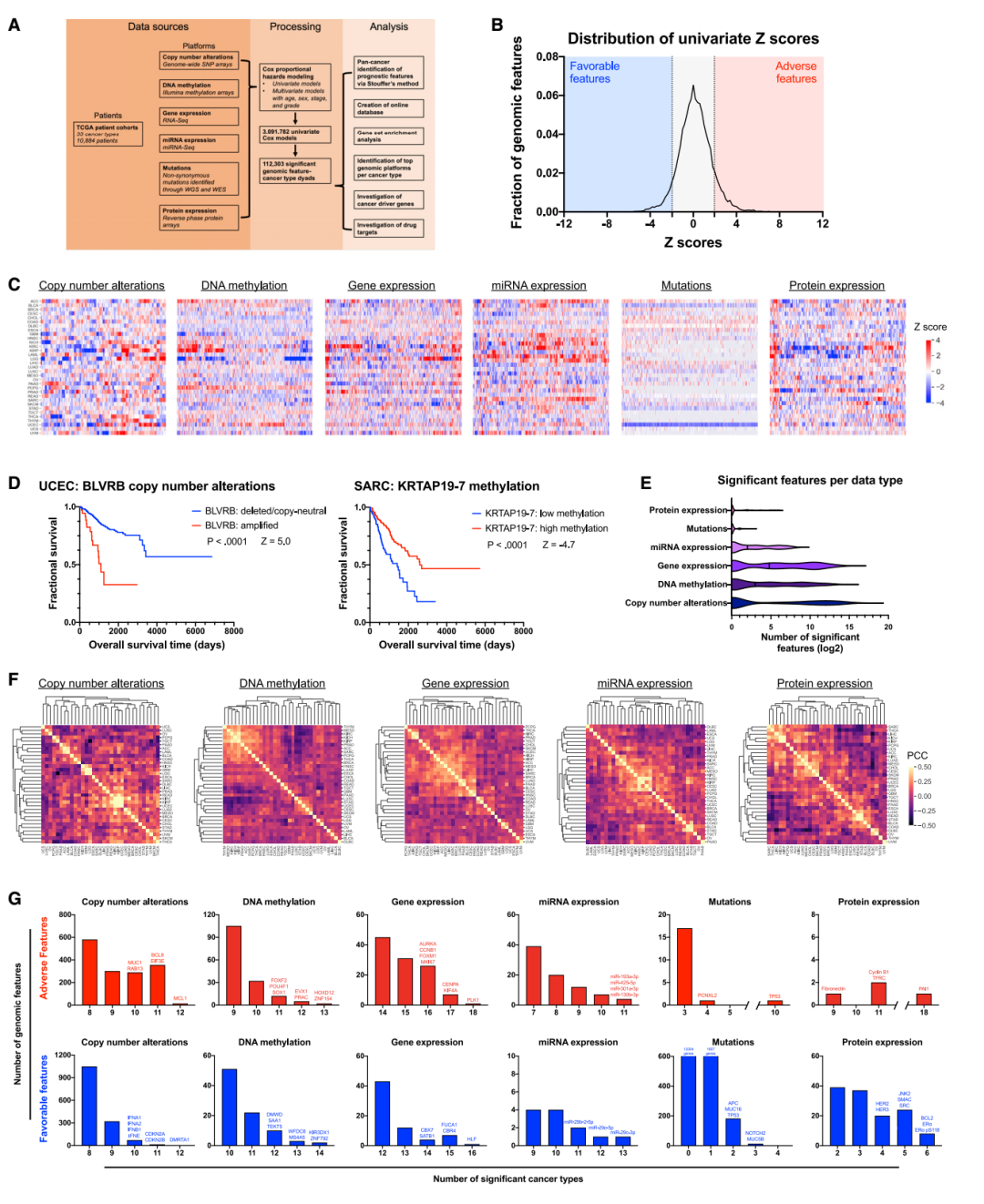
(A) 为这项工作进行的数据处理和分析的示意图。
(B) 显示所有六个平台(CNA、甲基化、突变、基因表达、miRNA 表达和蛋白质表达)的基因组特征 Z 分数分布的密度图。 Z = 1.96 处的虚线对应于有利特征的 p <0.05,而 Z = 1.96 处的虚线对应于
对于不利特征,p < 0.05。
(C) 显示六个基因组平台中 Z 分数分布的热图。每行对应一种癌症类型,每列对应一种基因或基因组特征。完整的 Z 分数集包含在表 S1 中。
(D) Kaplan-Meier 图显示了从我们的全基因组 Cox 模型中确定的两个具有代表性的预后生物标志物。 BLVRB 的拷贝数扩增与 UCEC 中较短的存活时间相关(左)。 KRTAP19-7 的甲基化与 SARC 中较长的存活时间相关(右)。
(E) 小提琴图显示每个基因组平台每种癌症类型的显着预后特征 (|Z| > 1.96) 的分布。
(F) 跨癌症类型的每个基因组平台的 Z 分数相关性的聚类图。尺度表示 Z 得分向量之间的 Pearson 相关系数的强度。
(G) 直方图显示跨癌症类型的每个基因组平台的共享预后生物标志物的数量
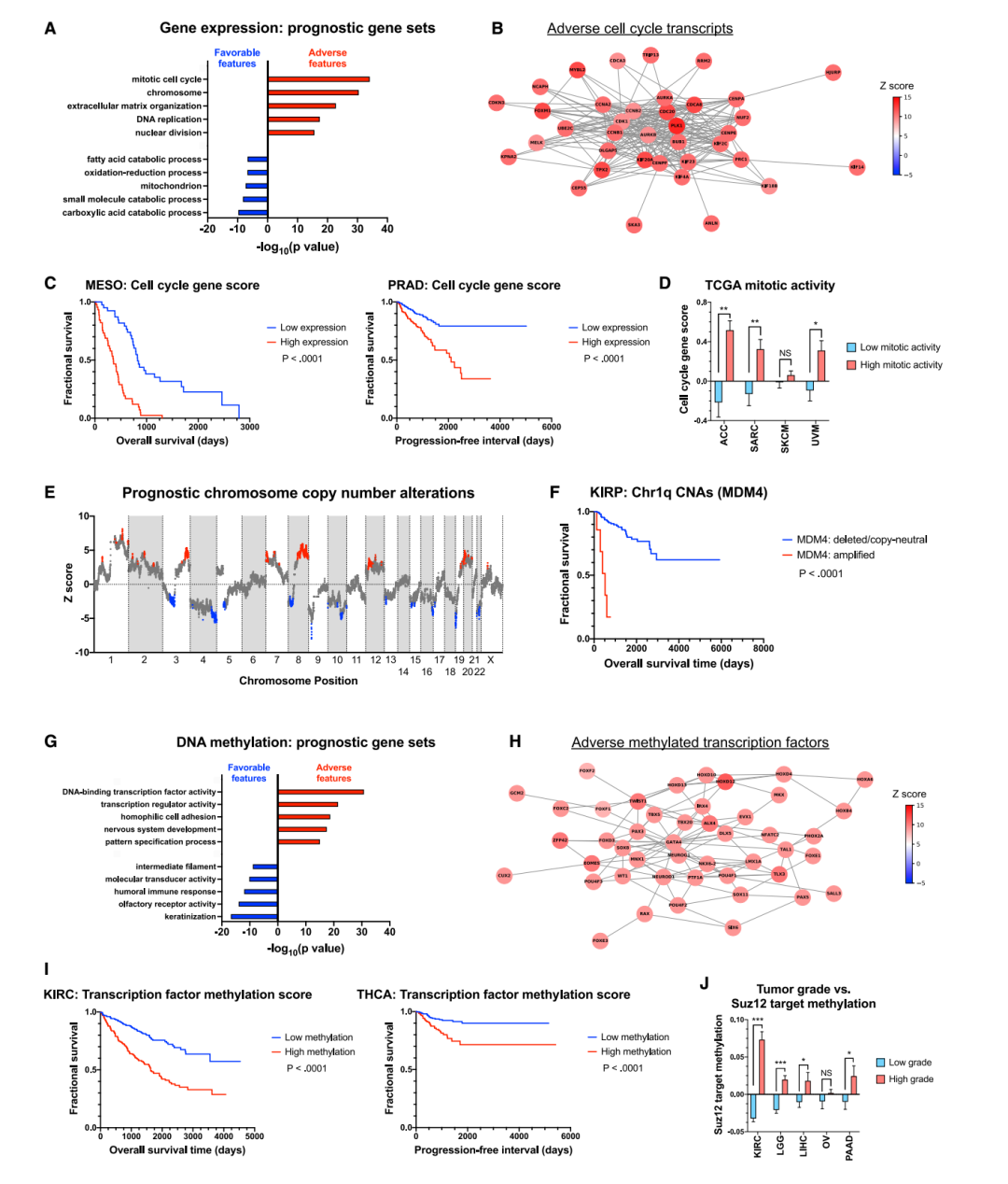
(A) GO术语在不利和有利的基因表达生物标志物中富集。完整的GO术语集包含在表 S4A 和S4B中。
(B) 细胞周期基因之间的相互作用网络,根据组合基因表达 Cox 模型的 Stouffer 的 Z 着色。
(C)MESO(左)和 PRAD(右)中的存活率的 Kaplan-Meier图,基于与基因本体术语“有丝分裂细胞周期”相关的一组转录本的平均表达。
(D) 条形图显示基于在不同 TCGA 队列中病理观察到的有丝分裂活性的细胞周期评分。
(E) 通过染色体坐标显示 Stouffer Z 的图。红点表示基因组扩增与较差结果相关的基因,蓝点表示基因组缺失与较差结果相关的基因。在这些区域中发现的完整基因列表包含在表 S5 中。
(F) Kaplan-Meier 图,根据 Chr1q 基因 MDM4 的拷贝数状态显示 KIRP 分裂中的存活率。
(G) GO 术语在不利和有利的甲基化生物标志物中富集。完整的 GO 术语集包含在表 S4E 和 S4F 中。
(H) 根据联合甲基化 Cox 模型的 Stouffer 的 Z 着色的发育转录因子之间的相互作用网络。
(I) Kaplan-Meier 图显示基于发育转录因子集合的甲基化在 KIRC(左)和 THCA(右)中的存活率。
(J) 条形图显示 Suz12 靶标在不同 TCGA 队列中基于肿瘤分级的平均甲基化。
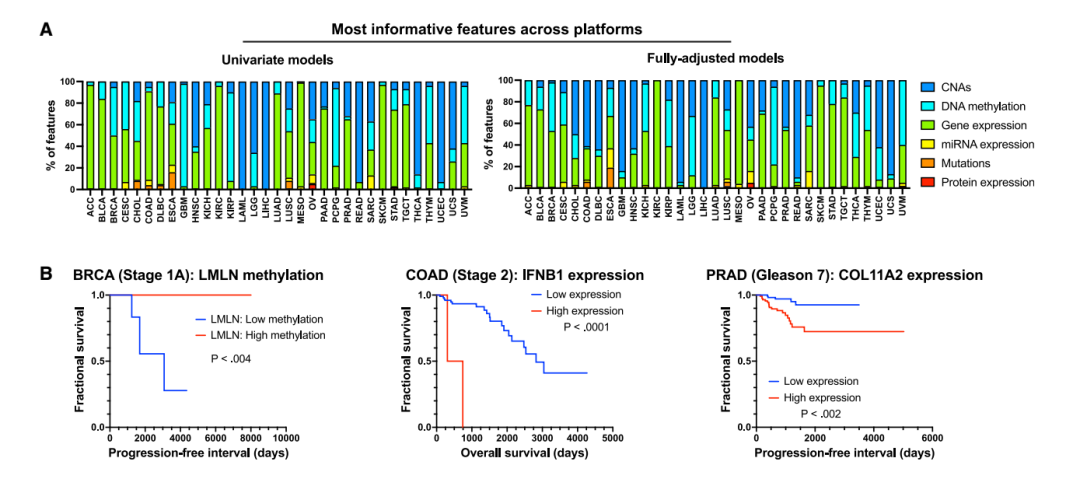
Fig. 3 跨平台分析显示癌症基因表达测量包含最有预后信息
(A) 显示在单变量或完全调整的 Cox 模型中与患者预后最相关的 100 个基因组特征。(B) Kaplan-Meier 图显示 1A 期乳腺癌、2 期结肠癌和 Gleason 7 期前列腺癌的患者存活率,根据指示的生物标志物进行分割。
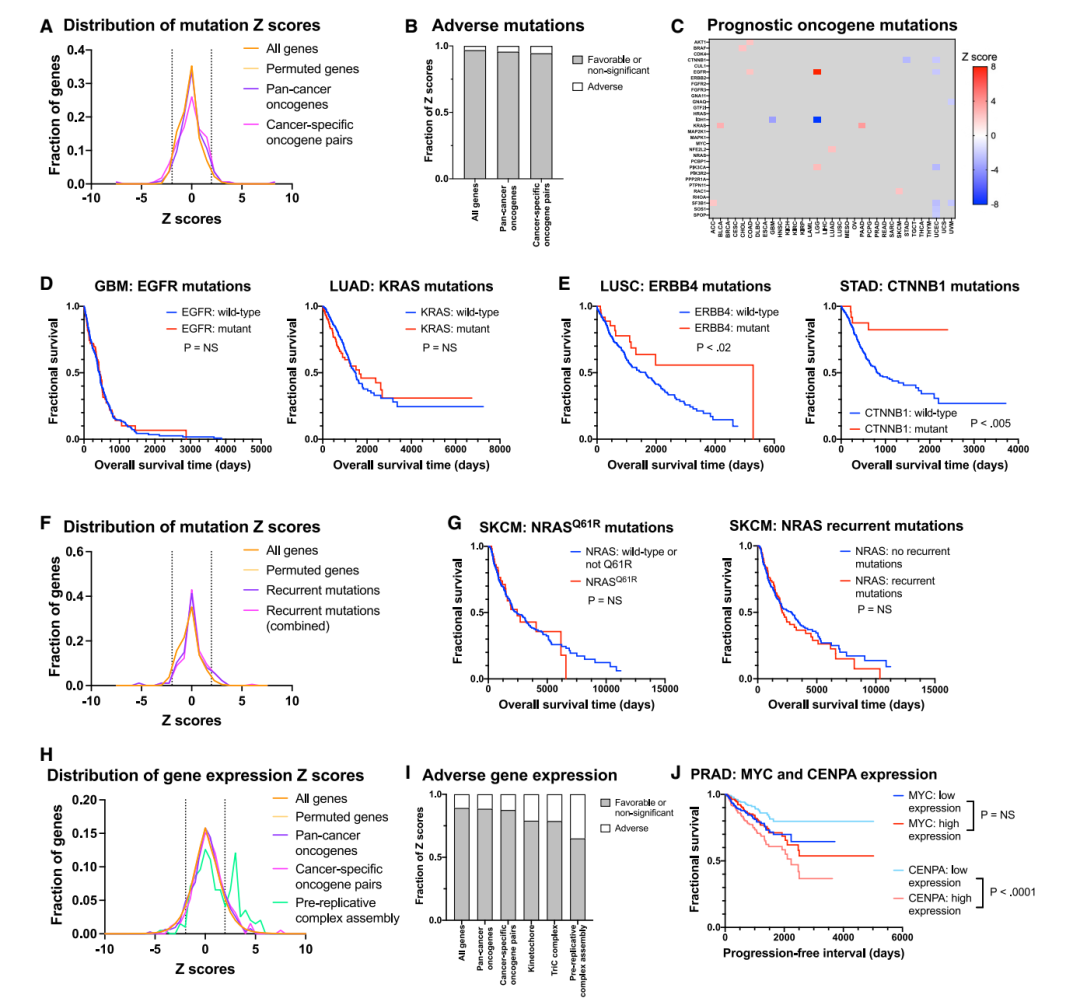
(A) 显示指定基因集的突变 Z 分数分布的密度图。 Z = 1.96 处的虚线对应于有利突变的 p < 0.05,而 Z = 1.96 处的虚线对应于不利突变的 p < 0.05。
(B) 堆叠条形图显示与指定基因组的不良结果相关的突变比例。
(C) 显示致癌基因突变的显着 (|Z| > 1.96) 生存关联的热图。
(D) Kaplan-Meier 图显示已建立的 GBM 癌基因 EGFR(左)和已建立的 LUAD 癌基因 KRAS(右)的突变与较短的存活时间无关。
(E) Kaplan-Meier 图显示 LUSC 癌基因 ERBB4(左)和 STAD 癌基因 CTNNB1(右)的突变与更长的存活时间相关。
(F) 显示指定基因组突变 Z 分数分布的密度图,包括影响特定反复突变密码子的“热点”突变。突变 Z 分数的完整列表包含在表 S6 中。
(G) Kaplan-Meier 图表明最常见的 NRAS 驱动突变 - Q61R - 或所有常见 NRAS 驱动突变的组合- G12D、G12S、G13D、G13R、K16N、Q61H、Q61K、Q61R和 E62K-不相关在 SKCM 中存活时间更短。
(H) 显示指定基因集的基因表达 Z 分数分布的密度图。请注意,虽然癌基因组与对照基因组在很大程度上重叠,但复制前的复杂组装基因组在对照基因组的右侧显示出显着的峰值。
(I) 堆叠条形图显示与指定基因组的不良结果相关的基因表达生物标志物的比例。
(J) Kaplan-Meier 图显示 PRAD 中的存活率基于关键驱动癌基因 (MYC) 的表达或基于非致癌细胞周期基因 (CENPA) 的表达的分裂。
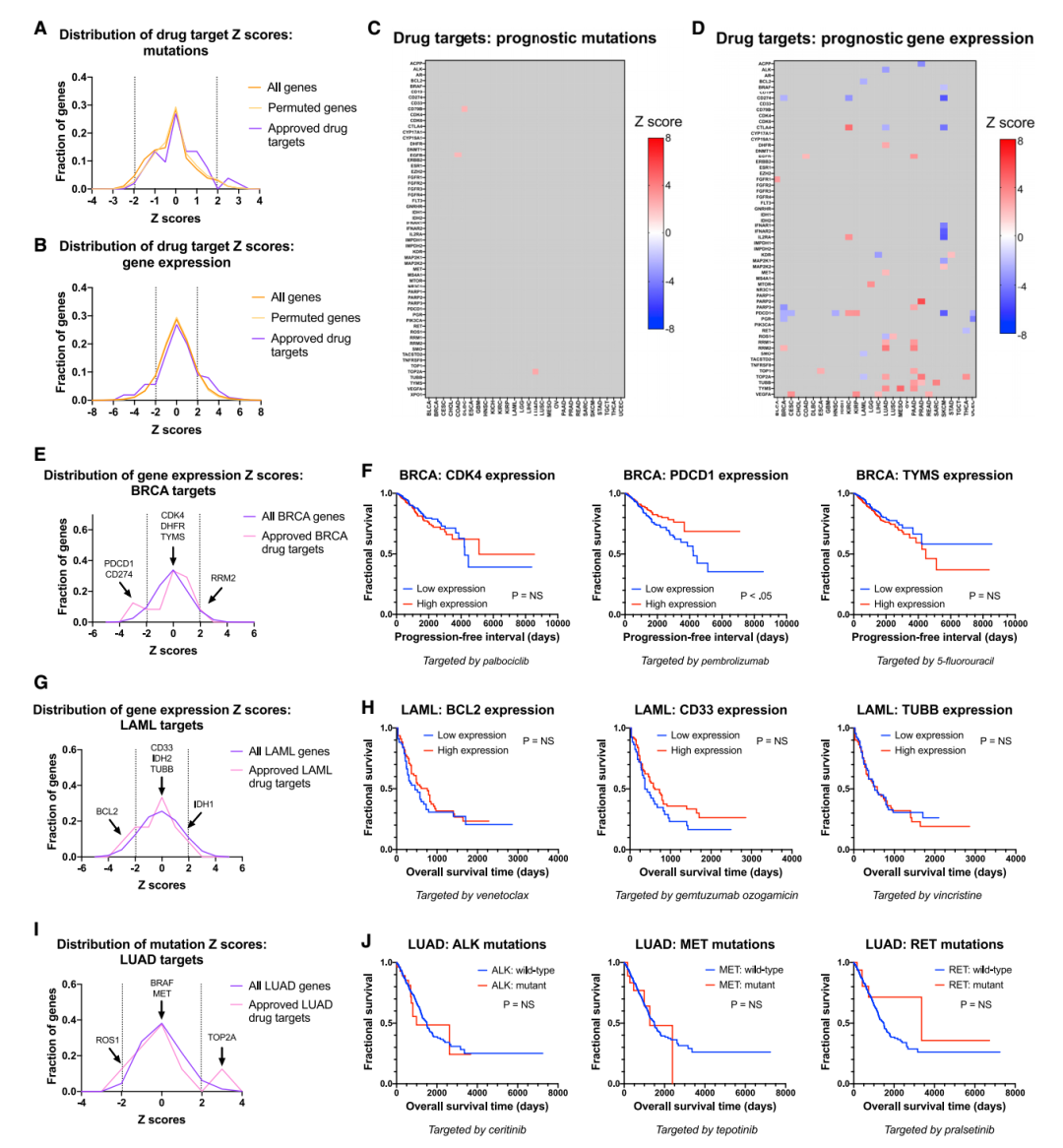
Fig. 5 FDA 批准的抗癌药物的基因组靶点并不是强有力的预后生物标志物
(A) 显示指定基因集的突变 Z 分数分布的密度图。 Z = 1.96 处的虚线对应于有利突变的 p < 0.05,而 Z = 1.96 处的虚线对应于不利突变的 p < 0.05。(B) 显示指定基因集的基因表达 Z 分数分布的密度图。
(C) 热图显示 FDA 批准的药物靶标突变与生存相关显着 (|Z| > 1.96)。 每行代表一个药物靶点,每列代表来自 TCGA 的癌症患者队列。
(D) 显示 FDA 批准药物靶点表达变化的显着 (|Z| > 1.96) 生存关联的热图。 每行代表一个药物靶点,每列代表来自 TCGA 的癌症患者队列。
(E) 密度图显示 BRCA 中指定基因集的基因表达 Z 分数分布。
(F) Kaplan-Meier 图根据 BRCA 药物靶向 CDK4、PCDC1 和 TYMS 的表达水平显示 BRCA 中的存活时间。
(G) 密度图显示 LAML 中指定基因集的基因表达 Z 分数分布。
(H) Kaplan-Meier 图根据 LAML 药物靶向 BCL2、CD33 和 TUBB 的表达水平显示 LAML 中的存活时间。
(I) 显示 LUAD 中指定基因集的突变 Z 分数分布的密度图。
(J) Kaplan-Meier 图根据指示的 LUAD 药物靶标 ALK、MET 和 RET 中的突变显示 LUAD 中的存活时间。
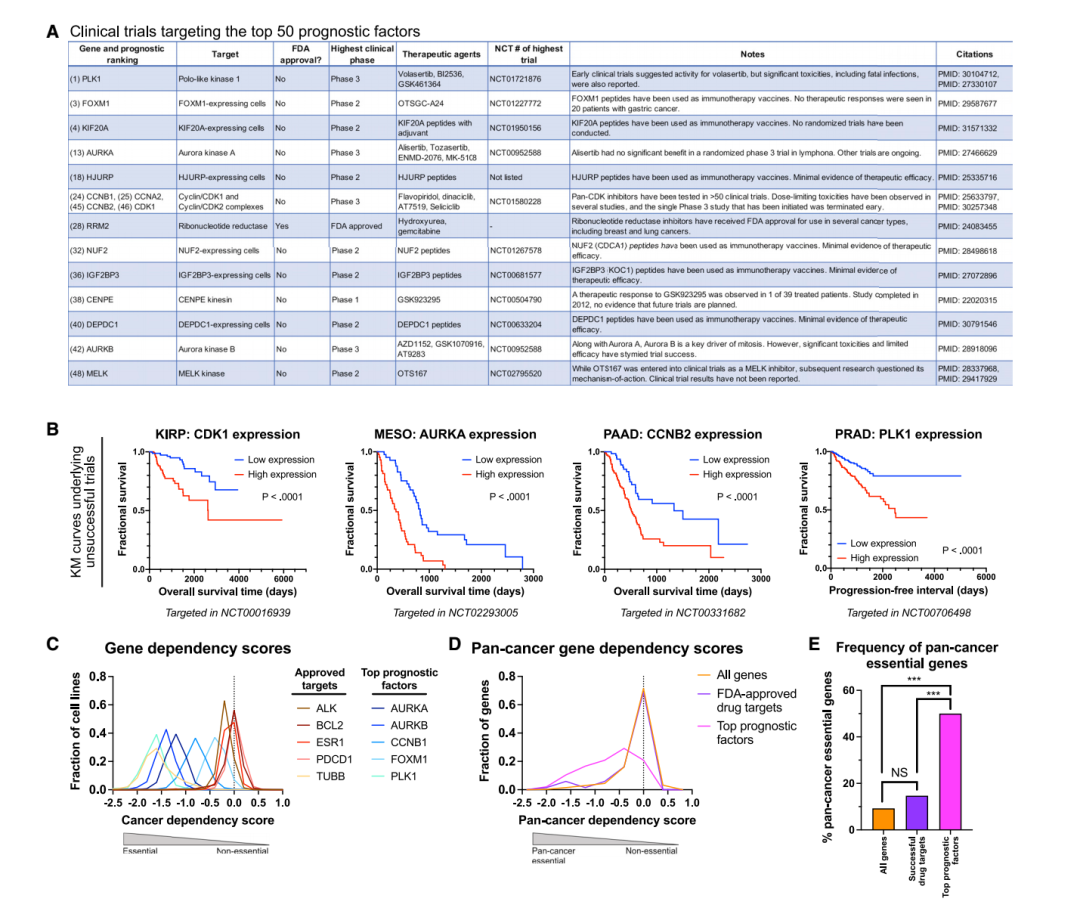
(A) 一张表格显示了 50 个预后因素中的基因,这些因素与癌症临床试验中针对的癌症患者结果表现出最强的相关性。
(B) Kaplan-Meier 图显示了指定癌症队列中的患者存活率。 每个图表都显示了一个基因,该基因已在该癌症类型的临床试验中被靶向。
(C) 显示指定基因的癌症依赖性评分分布的密度图,根据该基因是 FDA 批准的癌症治疗的靶标还是该基因是得分最高的预后因素进行划分。
(D) 显示指定基因组的泛癌癌症依赖性评分分布的密度图。
(E) 条形图显示在指定基因集中跨癌症类型必不可少的基因百分比。
生存时间较短的癌症特征并富含致癌基因或能成为好的药物靶点。
最强的不良生物标志物代表了广泛表达的细胞周期和管家基因。
研究为预后生物标志物分析建立了丰富的资源,并阐明了患者生存数据在临床前癌症研究和治疗开发中的用途。

