Cell Host & Microbe | 宿主细胞将营养生态位细化为肠道微生物离散的生物地理微生境
英文题目:Host cells subdivide nutrient niches into discrete biogeographical microhabitats for gut microbes
中文题目:宿主细胞将营养生态位细化为肠道微生物离散的生物地理微生境
期刊:Cell Host & Microbe
IF:21.023
DOI:https://doi.org/10.1016/j.chom.2022.04.012
1- 文章亮点
共生大肠杆菌利用肠腔上皮衍生的硝酸盐
带毒力沙门氏菌吸引吞噬细胞作为主要管腔硝酸盐来源;
缺乏毒力因子的沙门氏菌无法获得上皮衍生的硝酸盐
大肠杆菌利用吞噬细胞衍生的硝酸盐抑制沙门氏菌的生长
2- 文章概括
Liou等人发现致病性沙门氏菌和共生大肠杆菌在肠道腔内不同空间微生境中获得硝酸盐,这些空间微生境由不同的宿主细胞类型产生。大肠杆菌入侵沙门氏菌占据的空间微生境的能力有助于抗定植。
3- 研究背景
结肠拥有人体内最大的微生物群落,包含一百多种可以稳定维持数月的细菌。结肠栖息地包含 100 多个离散的营养生态位,但它们的特征仍然不明确。更好地了解饮食和宿主环境如何划分出每个可用的营养生态位对于充分理解微生物群的组装和功能至关重要。
膳食纤维的发酵是栖息地斑块中最成功的代谢特征之一。虽然膳食纤维在维持肠道微生物群中的优势细菌类群中的作用是一个深入研究的主题,但对少数物种占据的营养生态位知之甚少。
微生物群组成的变化与许多人类疾病有关,但控制菌株丰度的因素仍不清楚,因此分析控制菌株植入和丰度的因素与了解潜在的疾病过程相关。
4- 技术路线
构建大肠杆菌突变体→构建鼠伤寒沙门氏菌突变体→噬菌体转导→硝酸盐测定→构建骨髓嵌合体→流式细胞→组织处理→提取RNA和定量实时PCR→组织病理学→检测骨髓过氧化物酶→荧光成像→分割免疫荧光图像
Fig. 1 链霉素预处理小鼠结肠中大肠杆菌和鼠伤寒沙门氏菌微生境的可视化
(A-C)链霉素预处理的 lys-EGFP-ki 小鼠组感染了(A)鼠伤寒沙门氏菌野生型(STm WT),(B )一种无毒的 invA spiB 突变体(STm invA spiB),或(C)模拟感染 (Mock)。
(D-G)链霉素预处理的 C57BL/6 小鼠组(N 在 G 中表示)被模拟感染(模拟)或感染无毒 S. Typhimurium invA spiB 突变体(STm invA spiB),一种非运动的无毒 S. Typhimurium invA spiB fliC fljB 突变体 (STm invA spiB fliC fljB)、大肠杆菌 Nissle 1917 (EcN WT) 或非运动大肠杆菌 Nissle 1917 fliC 突变体 (EcN fliC)。
(D) 结肠切片对肌动蛋白(红色荧光)、DAPI 核染色(蓝色荧光)和鼠伤寒沙门氏菌 LPS 的 O12 抗原(白色荧光,上图)或大肠杆菌的 O6 抗原进行染色。大肠杆菌 Nissle 1917 LPS(白色荧光,底部面板)。
(E-G)定量细菌与上皮的距离。
(E) 通过分割肌动蛋白刷边界 (肌动蛋白边界) 和细菌定植区 (右上图) 对免疫荧光图像 (左上图) 进行定量分析的示例, 去除遮蔽区域 (左下图) 外的背景并生成种子 定殖区域内的像素代表细菌位置(右下图)
(F) 将每个种子像素与肌动蛋白边界内最近的像素(左面板)连接起来的线根据下面显示的配色方案进行着色以指示它们的长度。右图绘制了感染大肠杆菌 Nissle 1917 (EcN WT) 的小鼠切片的线数(值)与其长度。
(G) 该图绘制了感染无毒鼠伤寒沙门氏菌 invA spiB 突变体 (STm invA spiB) 的小鼠切片的线数 (值) 与其长度的关系。
(H) 该图显示了所示细菌菌株 (条) 与肌动蛋白边界距离的平均四分位数范围。
Fig. 2 大肠杆菌和鼠伤寒杆菌利用运动和能量趋向性在不同的微生境中获取硝酸盐
(A 和 B) 链霉素预处理的 C57BL/6 小鼠组 (N 由点数表示) 感染了大肠杆菌 Nissle 1917 (EcN) 或鼠伤寒沙门氏菌 (STm) 菌株和结肠内容物的指定混合物 感染后 6 天 (A) 或感染后 4 天 (B) 收集以确定竞争指数。
Fig.3 吞噬细胞衍生的硝酸盐构建了鼠伤寒沙门氏菌的营养生态位,共生大肠杆菌也可以获取该生态位
(A-D)链霉素预处理的 C57BL/6J 小鼠组被模拟感染(模拟)或感染大肠杆菌 Nissle 1917(EcN WT)、无毒的鼠伤寒沙门氏菌(STm invA spiB)或毒力的鼠伤寒沙门氏菌(STm WT)和器官在感染后 4 天收集。
(A) 盲肠的组织切片由兽医病理学家蒙蔽并评分。每个条形代表来自一种动物的数据。
(B) 指示的促炎基因的转录水平通过从盲肠粘膜分离的 RNA 中的定量实时 PCR 确定。
(C 和 D)每个点代表来自一只动物的数据。
(C) 通过改进的 Griess 测定法测定的盲肠粘液刮屑中的硝酸盐浓度。
(D) 通过 ELISA 测定盲肠内容物中髓过氧化物酶的水平。
(E 和 F) 致死照射的 C57BL6/J-Ly5.1 小鼠接受了来自野生型 (WT) C57BL/6 供体小鼠或 Nos2 缺陷供体小鼠的骨髓移植。
所得骨髓嵌合体小鼠组以及 C57BL/6 和 Nos2 缺陷供体小鼠组用链霉素预处理,然后用指定的鼠伤寒沙门氏菌菌株混合物感染。
(E) 从结肠内容物中回收的鼠伤寒沙门氏菌菌株的竞争指数。
(F) 显示来自骨髓嵌合体小鼠的肠细胞的流式细胞术分析的代表性图像,以区分受体的造血细胞 (CD45.1+) 与 C57BL/6- (上图) 或 Nos2 缺陷型 (下图) 的造血细胞面板)供体(CD45.2+)。
(G) 用指定的鼠伤寒沙门氏菌 (STm) 菌株感染链霉素预处理的 C57BL/6 小鼠组 (N 由点数表示),然后接种大肠杆菌 (EcN) 菌株混合物。
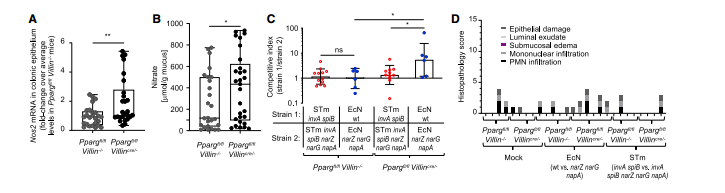
Fig. 4 上皮衍生的硝酸盐为大肠杆菌构建了一个营养生态位,无毒的鼠伤寒沙门氏菌无法进入
(A 和 B) 结肠上皮 (A) 或盲肠粘液 (B) 是从肠上皮 (Ppargfl/fl Villincre/) 或同窝对照 (Ppargfl/fl Villin/) 中缺乏 Pparg 表达的小鼠组中收集的。
(A) Nos2 转录水平是通过从结肠上皮制备物中分离的 RNA 中的定量实时 PCR 确定的。
(B) 粘液刮屑中的硝酸盐浓度通过改进的 Griess 测定法测定。
(A 和 B) 晶须印迹中的方框代表第一到第三个四分位数,线表示中值。每个点代表来自一种动物的数据。
(C和D)用指定的鼠伤寒沙门氏菌(STm)或E.大肠杆菌 (EcN) 菌株混合物。
(C) 感染后 4 天测定结肠内容物的竞争指数。
(D) 盲肠的组织切片由兽医病理学家蒙蔽并评分。

Fig. 5 McpB 和 McpC 将鼠伤寒沙门氏菌从含有营养的硝酸盐中排除
(A) 大肠杆菌 Nissle 1917(左图)和鼠伤寒沙门氏菌菌株 ATCC14028(右图)中趋化性受体基因(蓝色和红色箭头)的遗传库。
(B) 用指定的无毒鼠伤寒沙门氏菌 (STm) 菌株混合物感染链霉素预处理的 C57BL/6J 小鼠组,并在感染后 6 天收集盲肠内容物以确定竞争指数。
(C) 链霉素治疗的小鼠组感染了一种指定的鼠伤寒沙门氏菌菌株,2 天后收集盲肠进行切片。
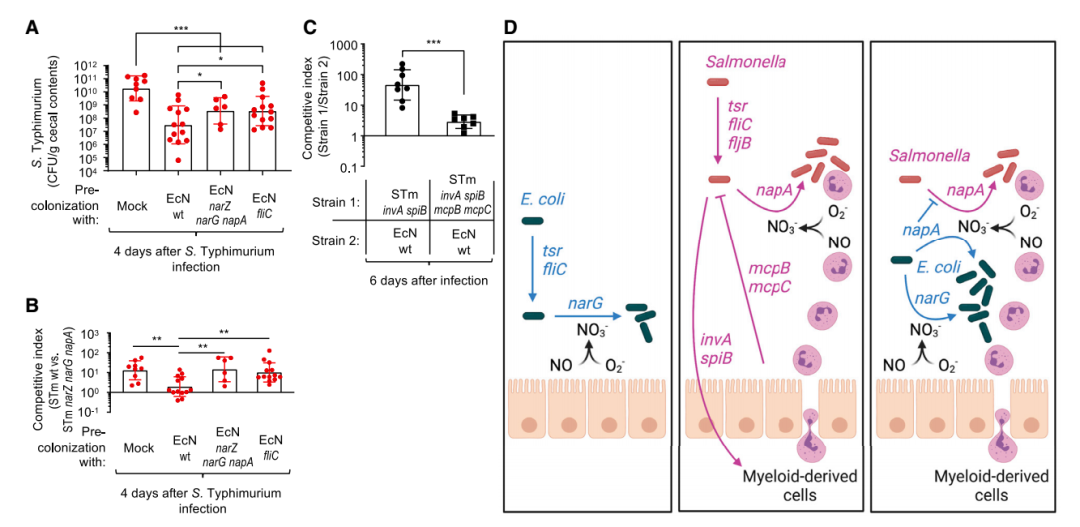
Fig. 6 大肠杆菌利用硝酸盐呼吸作用赋予对沙门氏菌的定植抗性
(A 和 B) 用无菌 LB 肉汤 (Mock) 或指定的大肠杆菌 Nissle 1917 (EcN) 菌株接种链霉素预处理的 C57BL/6J 小鼠组。接种大肠杆菌两天后,小鼠受到鼠伤寒沙门氏菌 (STm) 野生型 (WT) 和硝酸盐呼吸缺陷突变体 (narZ narG napA) 的 1:1 混合物的攻击。
(A) 从盲肠内容物中回收的鼠伤寒沙门氏菌总数。
(B) S. Typhimurium 菌株的竞争指数。
(C) 用指定的鼠伤寒沙门氏菌和大肠杆菌菌株的混合物接种链霉素预处理的 C57BL/6J 小鼠组。该图显示了比较鼠伤寒沙门氏菌菌株与大肠杆菌菌株的适应性的竞争指数。
(D) 共生大肠杆菌(左图)、毒性鼠伤寒沙门氏菌(中图)和两种物种之间对硝酸盐的竞争(右图)所占据的营养生态位模型。
5- 研究结果
1. 一种共生大肠杆菌菌株和一种致病性肠沙门氏菌血清型鼠伤寒分离株都利用硝酸盐促进肠道生长,它们在不同的生态位环境中获取这种资源。
2. 共生大肠杆菌利用上皮衍生的硝酸盐,而鼠伤寒沙门氏菌占据的生态位中的硝酸盐则来自吞噬细胞浸润。
3. 无毒鼠伤寒沙门氏菌不能利用上皮衍生的硝酸盐,因为其趋化受体 McpB 和 McpC 将病原体排除在大肠杆菌占据的生态位之外。
4. 大肠杆菌侵入由鼠伤寒沙门氏菌毒力因子构建的生态位,并通过竞争硝酸盐赋予定殖抗性。
5. 营养生态位不仅仅由关键资源定义,它们还可以在宿主内进一步细分为不同的微生境,从而为不同的细菌物种创造新的生态位机会。

